Konwencjonalne testy identyfikacji bakterii zazwyczaj wymagają kilku godzin na amplifikację i hybrydyzację DNA, godzin na reakcję przeciwciało-antygen lub nawet dni na hodowlę bakterii. W porównaniu z tymi metodami spektroskopia Ramana jest atrakcyjna do analizy składników chemicznych i biologicznych bez konieczności skomplikowanego przygotowania próbek. Spektroskopia Ramana jest techniką spektroskopii optycznej opartą na nieelastycznie rozproszonym świetle. Powstałe widmo Ramana stanowi „molekularny odcisk palca” próbki, dostarczając wysoce szczegółowych informacji o składzie chemicznym i strukturze molekularnej. W przypadku identyfikacji bakterii widmo Ramana dostarcza informacji o składzie chemicznym komórek, a także strukturze ich biomolekuł, takich jak: białka, lipidy czy kwasy nukleinowe.
Specyficzność widma Ramana jest ogromna. Już od pierwszych doniesień o efekcie Ramana w 1928 r. aż, do początku lat 80. XX w. technika ta ze względu na stopień komplikacji niektórych komponentów, takich jak lasery, detektory, ograniczała się jedynie do akademickich badań laboratoryjnych. Chociaż ten rozwój spektroskopii Ramana stanowił znaczny postęp w stosunku do oryginalnego oprzyrządowania, układy do spektroskopii Ramana były nadal złożone, nieefektywne i trudne w obsłudze. Jedną z przyczyn tej trudności była nieefektywność i niestabilność elementów optycznych. Przełom nastąpił w latach 90. XX w., kiedy m.in. firma Renishaw wprowadziła na rynek prosty w obsłudze spektrometr inViaTM Raman wyposażony w mikroskop, co umożliwiło przejście tej techniki z pomiarów w makro do mikro skali. Od tego czasu dodatkowe ulepszenia elementów optycznych, stabilność laserów, automatyzacja, obrazowanie, kompaktowa konstrukcja spektrometru, możliwość łączenia z innymi technikami analitycznymi, jak mikroskopia sił atomowych (AFM/TERS), mikroskopia elektronowa (SEM) i światłowodowe sondy Ramana, jak w analizatorze VirsaTM Raman Renishaw, pomogły uczynić spektroskopię Ramana praktyczną technologią także poza laboratorium, co jest kluczowe w przypadku szybkich testów diagnostycznych czy też identyfikacji zagrożeń mikrobiologicznych w przemyśle spożywczym, bioprzetwarzaniu lub na polu walki.
Bakterie występują w różnych rozmiarach i kształtach. Ich rozmiar, jeżeli spojrzymy na rysunek poniżej (rys. 1), to 1/10 komórek eukariotycznych. Mają ok. 0.5-5.0 um długości. Wystarczy nadmienić, że najmniejsza zbadana bakteria – Mycoplasma genitalium – ma wielkość ok. 200-300 nm, czyli mieści się na granicy limitu dyfrakcji dla rozdzielczości konwencjonalnego mikroskopu Ramana. Pokazuje to, jak istotną kwestią jest odpowiedni dobór instrumentarium w przypadku badania bakterii – długości lasera, rozdzielczości przestrzennej i spektralnej spektrometru, a także obiektywu o odpowiednim powiększeniu. W celu uzyskania widm o większej intensywności często w badaniach bakterii używana jest metoda wzmocnionej powierzchniowo spektroskopii Ramana (SERS), w której stosuje się nanocząstki metaliczne (NPs) Au lub Ag bądź specjalnie przygotowane chropowate podłoża metaliczne, o różnej grubości warstwy Au.

Uzyskane widma bakterii, tak jak i innych próbek biologicznych, są bardzo złożone. Poniżej wymieniono charakterystyczne pasma oraz rodzaje wiązań, które mogą być identyfikowane przy pomocy spektroskopii Ramana:
• pasmo C-H – charakterystyczne pasmo związane z wiązaniami C-H w lipidach komórkowych, białkach i kwasach nukleinowych,
• pasmo Amide I – związane z wiązaniami C=O i N-H w białkach, co umożliwia analizę struktury białek w komórkach,
• pasmo Amide III – związane z wiązaniami N-H i C-N w białkach, stanowiące dodatkowe informacje o strukturze białkowej,
• pasma nukleinowe – charakterystyczne pasma związane z wiązaniami w kwasach nukleinowych, pozwalające na analizę DNA i RNA,
• pasmo CH2/CH3 (1454 cm-1) – związane z wiązaniami C-H w lipidach, co umożliwia analizę składu lipidowego komórek bakteryjnych,
• Pasmo peptydowe – pasmo związane z wiązaniami peptydowymi w białkach, co pozwala na identyfikację i analizę struktury peptydów,
• pasmo fosfolipidowe – związane z fosfolipidami, umożliwiające analizę składu lipidowego błon komórkowych,
• pasma DNA/RNA – charakterystyczne pasma związane z wiązaniami fosforanowymi w DNA/RNA, co pozwala na analizę kwasów nukleinowych,
• pasmo Amide II – związane z wiązaniami N-H i C-N w białkach, wspierające analizę struktury białek.
Zwykle bakterie są dla nas nieszkodliwe, jednak niektóre szczepy, takie jak np. S. aureus czy Bacillus cereus, mogą powodować poważniejsze infekcje, jeśli przedostaną się do organizmu. Postępowanie z pacjentami z infekcjami bakteryjnymi opiera się na wczesnym wykrywaniu i identyfikacji patogenów, gdyż umożliwia to odpowiednie podanie antybiotyków, co często ratuje życie. W przypadku poważniejszych schorzeń, np. posocznicy, leczenie należy rozpocząć w ciągu godziny od rozpoznania. Niestety, wykonanie obecnych testów, jak już wspomnieliśmy, często zajmuje kilka dni, co może narażać życie pacjenta na niebezpieczeństwo. Stąd możliwość wykorzystania spektroskopii Raman do szybkiej identyfikacji bakterii jest tak kluczowe dla rozwoju szybkiej diagnostyki. Takie badanie może obejmować następujące aspekty:
• identyfikację gatunku – analiza widm Ramana pozwala na identyfikację charakterystycznych sygnałów związanych z danym gatunkiem bakterii; w przypadku Bacillus cereus można znaleźć pasma charakterystyczne dla tej bakterii,
• skład chemiczny komórek – spektroskopia Ramana pozwala na analizę składu chemicznego komórek bakterii, obejmując białka, lipidy, kwasy nukleinowe i inne składniki; to umożliwia szczegółową charakteryzację mikroorganizmu,
• badanie morfologii komórek – analiza widm Ramana może dostarczać informacji na temat morfologii komórek bakterii, takich jak obecność otoczki czy struktury wewnątrzkomórkowe,
• ocenę stanu metabolicznego – Raman może dostarczać informacji na temat stanu metabolicznego bakterii, co może być istotne w badaniach nad reakcjami komórkowymi,
• wykrywanie zmian wzrostu i warunków środowiskowych – badania Ramana mogą być stosowane do monitorowania zmian w komórkach bakterii w zależności od warunków wzrostu i środowiska.
Zobaczmy, jak spektroskopia Ramana sprawdzi się na przykładzie wspomnianej już bakterii Bacillus cereus. Na rysunku poniżej (rys. 2) widzimy obraz mikroskopowy Bacillus cereus i nałożoną na niego mapą Ramana. Zmiany obserwowane na załączonych poniżej widmach wynikają z różnicy stanu uwodnienia rdzenia zarodników bakterii, o czym świadczą zaznaczone na widmach zmiany intensywności pasm w zakresie od 1000 cm-1 do 1030 cm-1 dla kwasu Ca2+ - dipikolinowego (Ca-DPA), w tym dodatkowo widoczne są wyraźne pasma: 825 cm-1, 1017 cm-1, 1451 cm-1 i 1574 cm-1. Kwas Ca-DPA pełni kluczową funkcję w procesie sporulacji bakterii Gram-dodatnich, w szczególności dla rodzaju Bacillus, w tym Bacillus cereus, Bacillus anthracis i Bacillus subtilis. Ważne jest zauważenie, że kwas Ca-DPA jest specyficznym dla bakterii Gram-dodatnich i nie występuje powszechnie w innych typach bakterii, stąd może być w spektroskopii Ramana swoistym markerem do rozróżniania tych typów bakterii. Jego rola w sporulacji czyni go interesującym celem badań w kontekście mikrobiologii i biologii komórkowej. Dlatego właśnie określenie i śledzenie zmian stosunku pasm Ramana charakterystycznych dla Ca-DPA pozwala na szybkie wykrywanie bakterii Gram-dodatnich i określenia stopnia ich aktywności, uwodnienia, czyli przejścia w formę endospory.
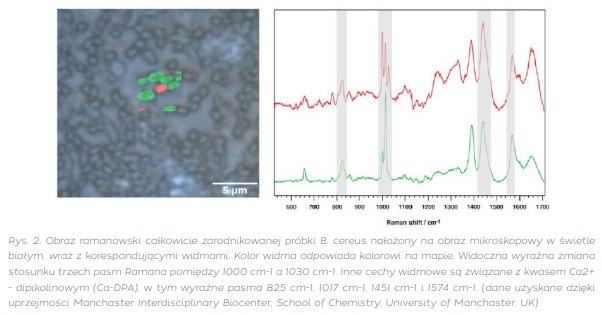
Ze względu na ten sam rodzaj, podobne rozmiary i cechy na widmie Ramana zarodniki Bacillus cereus stanowią użyteczny model do zrozumienia zachowania i wykrywania zarodników Bacillus anthracis – bardzo niebezpiecznej bakterii, która jest przyczyną choroby znanej jako wąglik (antraks). Warto zauważyć, że Bacillus anthracis był także używany jako broń biologiczna, co podkreśla wagę monitorowania tej bakterii i środków ostrożności wobec potencjalnego zagrożenia bioterrorystycznego. Szybka analiza setek tysięcy widm wymaga jednakże odpowiedniego oprogramowania, które posiada narzędzia do analizy statystycznej danych pomiarowych. Zaawansowane metody analizy statystycznej, takie jak analiza składowych głównych (PCA), częściowa metoda najmniejszych kwadratów (PLS) czy klasyfikacja przy użyciu algorytmów maszynowego uczenia, mogą być wykorzystywane do automatycznej identyfikacji bakterii na podstawie zmian cech w widmach Ramana. Ponadto możliwość wykonywania pomiarów Ramana także in situ, in vivo, czyli wyjścia poza strefę laboratorium, jest możliwe dzięki użyciu spektrometrów przenośnych wyposażonych w odpowiednie ramanowskie sondy światłowodowe, takie jak np. oferuje spektrometr VirsaTM Raman Renishaw (rys. 3).

Podsumujmy zatem korzyści, jakie daje nam zastosowanie spektroskopii Ramana w diagnostyce bakteryjnej. Są to:
• szybkość i precyzja,
• brak konieczności hodowli, możliwość pomiarów in vivo,
• wielogatunkowa analiza,
• możliwość analizy pobranych bezpośrednio próbek klinicznych,
• monitorowanie reakcji na leki i antybiotyki,
• automatyzacja procesu – integracja spektroskopii Ramana z technologią Artificial Inteligence (AI), maszynowego uczenia pozwala na automatyczne rozpoznawanie wzorców i skrócenie czasu analizy.
Spektroskopia Ramana wykazuje zarówno niezbędną ze względu na rozmiary bakterii rozdzielczość przestrzenną, jak i czułość chemiczną, aby pomóc w szybkiej identyfikacji bakterii. Otwiera nowe perspektywy w diagnostyce, monitorowaniu lekooporności oraz analizie składu chemicznego komórek bakteryjnych. Pozwala rozwikłać złożone procesy biologiczne, a także monitorować środowisko i produkty spożywcze pod kątem obecności bakterii i ich zarodników, co jest istotne dla bezpieczeństwa żywności i zdrowia ludzi.
Więcej informacji na temat zastosowań Ramana prod. Renishaw oraz przykładów ich zastosowań można znaleźć na stronie firmy. Dzięki bogatemu doświadczeniu w czołówce technologii przyrządów ramanowskich od ponad 30 lat, wspieranemu przez globalną strukturę, firma Renishaw jest partnerem, na którym można polegać teraz i w przyszłości. Zapraszamy do współpracy!
Autorka: Agnieszka Sozańska, Kierownik Sprzedaży Produktów Spektroskopowych w Renishaw





















KOMENTARZE